パリピをエッジ検出してみた。
最近は、ハウスメーカーとの打ち合わせで忙しいです。
いきなりのカミングアウトすみません。
パリピ女子をエッジ検出してみた。
今回使用するパリピ女子のフリー素材はこちら↓

(いい感じっすねぇー)
まずは、平滑化↓

簡単ver. ↓
import cv2
img = cv2.imread("paripi.jpg")
img_blur = cv2.blur(img, (3,3))
cv2.imshow("img",img_blur)
cv2.imshow("src",img)
cv2.waitKey(0)
cv2.destroyAllWindows()
少し、ボケた感じになります。
簡単にエッジ検出

エッジりすぎました。
img_canny = cv2.Canny(img, 5, 100)
cv2.imshow("Canny",img_canny)
cv2.waitKey(0)
cv2.destroyAllWindows()

少しエッジが減りました。
img_canny = cv2.Canny(img, 100, 200)
cv2.imshow("Canny",img_canny)
cv2.waitKey(0)
cv2.destroyAllWindows()
結論:パリピ女子はやっぱり普通の画像の方が可愛いですね。
そやけども、パリピ具合は検出できそうですね。
前回は、美女子をフーリエ変換したのですが、今回もパリピ女子をフーリエ変換してみました。

やっぱり、パリピ女子をフーリエ変換しても可愛くはないですね。w
from PIL import Image
import numpy as np
from matplotlib import pyplot as pltdef main():
img = Image.open('paripi.jpg')
gray_img = img.convert('L')
f_xy = np.asarray(gray_img)# 2 次元高速フーリエ変換で周波数領域の情報
f_uv = np.fft.fft2(f_xy)
# 画像の中心に低周波数の成分がくるように
shifted_f_uv = np.fft.fftshift(f_uv)# パワースペクトル
magnitude_spectrum2d = 20 * np.log(np.absolute(shifted_f_uv))
unshifted_f_uv = np.fft.fftshift(shifted_f_uv)
i_f_xy = np.fft.ifft2(unshifted_f_uv).real
fig, axes = plt.subplots(1, 3, figsize=(40, 20))
for ax in axes:
for spine in ax.spines.values():
spine.set_visible(False)
ax.set_xticks()
ax.set_yticks()
axes[0].imshow(f_xy, cmap='gray')
axes[0].set_title('Input Image')
# 周波数領域のパワースペクトル
axes[1].imshow(magnitude_spectrum2d, cmap='gray')
axes[1].set_title('Magnitude Spectrum')
axes[2].imshow(i_f_xy, cmap='gray')
axes[2].set_title('Reversed Image')
plt.show()
if __name__ == '__main__':
main()
今回も、しょうもない記事を見ていただいてありがとう!!
美人をフーリエ変換してみた。(空間周波数領域っていうのがあるんですよ。)
どうも、最近はモンハンで忙しいです。
いきなりのカミングアウトすみません。
美人のフリー画像をフーリエ変換してみた。
(空間周波数領域っていうのがあるんですよ。)
前回は、自分の音声をフーリエ変換しました。
今度は、美人のフリー画像をフーリエ変換してみました。
使用するが画像はこちら↓ (美人で検索して出てきたフリー画像のようです。)

(確かに美人ですね。w)
人間が直接に見ることのできない現象や事象を画・グラフ・図などにすることを可視化 : Visualizationと呼びますが、まぁそんな方法の一つです。
いきなりですが、美人をフーリエ変換しました↓↓。Pythonでしました。
どうですか↓↓? やっぱり美人はフーリエ変換しても美人ですね。

なんか、すみません。画像をフーリエ変換したら、人ではなくなりました。
検証結果:美人をフーリエ変換すると人ではなくなる。
(当たり前の結果ですみません。)
今回もしょうもない記事を読んでいただいてありがとうございます。
自分の声(’寿司食いてぇ’)をフーリエ変換してみた。
どうも、右の拇指球にタコができてしまいました。
いきなりのカミングアウトすみません。
自分の声をフーリエ変換。
フーリエ変換とは、、なんぞや。下の数式は公式みたいよ。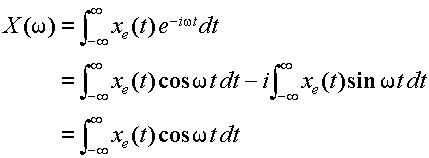
(これは、高速フーリエ変換です。すみません)
とにかく、フーリエ変換というのは、時間tの関数から角周波数ωの関数への変換です。(難しい話は、また今度)
音の波形を分かりやすくしたものがフーリエ変換です。スペクトルとか周波数とか、難しい言葉出てきたり、サインとコサインが出てきたり、微分積分が好きな人は、フーリエ勉強してみてください。
今回も、使用した音声は「寿司食いてぇ」です。
Dropbox - Sushi.m4a - Simplify your life
使用したコード↓
import sys
import scipy.io.wavfile
import numpy as np
import matplotlib.pyplot as plt
#音声ファイル読み込み
args = sys.argv
wav_filename = args[1]
rate, data = scipy.io.wavfile.read('Pap.wav')
#(振幅)の配列を作成
data = data / 32768
#時間領域から周波数領域に変換
fft_data = np.abs(np.fft.fft(data))
#横軸:周波数取得
freqList = np.fft.fftfreq(data.shape[0], d=1.0/rate)
#データプロット
plt.plot(freqList, fft_data)
plt.xlim(0, 4000)
plt.show()

今回もしょうも無い記事ですみません。
Pythonでサウンドスペクトログラムを作ってみた。
どうも、日本酒ロスのグチナカです。
いきなりのカミングアウトすみません。
最近は音声処理や解析をマイブームにしております。(実は動画m)
音声の分類問題や特徴量を、、、、云々と勉強しておりますが、なかなか難しいです、たぶん。
Pythonでスペクトログラムを作ってみた。
ということで、スペクトログラムを作ってみました。
スペクトログラムってなんぞや!?はい、言語聴覚士なら知っているみたいですよ。普通は知らないですよね。
音声には振動があります。振動の波形と時間を可視化したものはこちら↓

上記の画像は、
「お寿司たべたーいなっ!」という言葉を可視化したものです。
(出来れば回らないシースーがくいてぇ、言葉のチョイスについては謝罪致します。)
上記画像は、たまに見かける事がありそうです。単純な音声の可視化なので、iPhoneならボイスレコーダーで録音する時にも確認できます。
スペクトログラムは、横軸が時間で縦軸が周波数になります。そして、色で周波数の強度を表します。
お寿司を食べたいの音声を一度聞いてみて下さい。↓↓
Dropbox - Sushi.m4a - Simplify your life
「お寿司たべたーいなっ!」のスペクトログラムはこちら↓↓

まぁ、こんな感じですよ。
Twitterのテキストデータを感情分析:感情スコアを出す。 - 人工知能・リハビリ・日記
私のリハノメの見方; Fire stick + アンドロイドでテレビに無線で出力する!
リハビリ関連職のE-ラーニング ”リハノメ”ですが、PCで見るのもよし、スマホで見るのもよしですが、やっぱり大画面で見たい!!
リハノメ | [株式会社gene]コメディカル向けセミナーと介護保険事業・出版事業
でも、HDMIで繋ぐのは面倒くさいし、、、。と思っていたのですが、Amazon Fire TV stickとアンドロイドのスマホがあれば1分で、大画面のテレビで見ることが出来ました!もちろん、無線です!!有益そうなので、共有いたします。
(iPhoneでも可能ですが、有料アプリが必要です。)
アマゾンFire stick とAndroidでテレビにリハノメを映す
準備は3つ !!
1. Fire TV stick
3. 1と2を同じwifiに繋いでおく
準備が出来たら手順はこちら↓
1. Fire stick のホームボタンを長押してミラーリングの準備
これだけです。
・Fire stickを起動した状態で、下の写真にあるホームボタンを長押してください。

・このような画面↓ がテレビに映し出されるので、ミラーリングを選択

次はスマホです。
・キャストボタンを押すだけ
(ミラーリングを許可していない方は許可をしてください)

もし、キャストがなければ、設定からキャストで検索してみてください。
Androidのスマホでキャストのボタンを押すと自動で接続が始まります!
後は、いつも通りスマホでリハノメを映すだけで、テレビの大画面で勉強できます!!!
こんな感じ!

うちでは、BGMのようにリハノメを流していることがあります(逆にすみません)。今日は、運動器の画像の見方をみました!!
F値は2つある?F statistics and F measure!

どうも、お酒は週4以下にしています。グチナカです。体脂肪率が8割を超えました。
いきなりのカミングアウトすみません。
F値は2つある? F statistics and F measure
この前、ある機会があり、F値の話になりました。F値くるぅーという感じで、よく使うと即答したのですが、F measureではなく、F statisticsでした👍
F statistics : F統計量
分類問題や予測精度の評価をする場合は、F measureを使用します。一方、F statisticsはF検定などで出てくるものです。F検定というのは、分散分析ですね。t検定のt値と解釈としては、同じですかね。
t値の場合は、まぁ自由度により変わりますが、α=0.05は、t値(z値)が1.96ですが、F検定は3群以上で使われる事が多いので、群などの自由度により、α=0.05に対するF statisticsは異なります。F分布表をみれば分かるので、見てみて下さい。
http://ktsc.cafe.coocan.jp/distributiontable.pdf
F measure
こちらのF値は、分類問題や診断の予測精度の指標です。0-1の範囲で、1が予測精度が高いという解釈です。これは、混同行列から算出される指標です。かなりざっくり説明すると、再現率と適合率の調和平均です。
混同行列については、また今度書きましょうか。
とりあえず、F値は2種類あります。
・分散分析(F検定)などで使用されるもの
→ F statistics : F統計量
・予測精度を表すもの
→ F measure : F メジャー
Decision Curve Analysis(DCA)について:意思決定曲線分析 - 人工知能・リハビリ・日記
Twitterのテキストデータを感情分析:感情スコアを出す。 - 人工知能・リハビリ・日記
画像を正規化:Pythonコード 記事は雑です。
どうも、最近、○○学という雑誌にニューラルネットワークを使った論文がアクセプトされました。カミングアウトすみません。
画像を正規化:Pythonコード
意味不明と思っているかたも多いかと思います。
正規化は機械学習をするにあたり重要な処理の一つです。PyTorchを使いました。
import torch
from torchvision import datasets, transforms
from torch.utils.data import DataLoader
import os
class calc_mean_std:
def __init__(self) :
print("calc_img")def calc(self, train_data,batch_size,num_workers):
data_transform = transforms.Compose([transforms.ToTensor()])
train_data.dataset.transform = data_transform
dataloader = DataLoader(train_data, batch_size=batch_size,
shuffle=False, num_workers=num_workers)
mean = 0.0
for images, _ in dataloader:
batch_samples = images.size(0)
images = images.view(batch_samples,images.size(1),-1)
mean += images.mean(2).sum(0)
mean = mean/len(dataloader.dataset)var = 0.0
for images, _ in dataloader:
batch_samples = images.size(0)
images = images.view(batch_samples,images.size(1),-1)
var += *1**2).sum([0,2])
std = torch.sqrt(var/(len(dataloader.dataset)*512*512))
print(mean,std)return mean,std
大体はコピペで使用出来ます。
*1:images - mean.unsqueeze(1
Decision curve analysisのR code:DCA、意思決定曲線分析
どうも、今回はタイトルそのままです。
データでコードを公開しても良いのですが、。
DCAとは?という方はこちらから読んでください↓
Decision Curve Analysis(DCA)について:意思決定曲線分析 - 人工知能・リハビリ・日記・理学療法
適当に作ったサンプルデータなので、こんな感じです。

Decision curve analysis:DCA R code
描画コードは2つに分けております。
描画↓
DCA
rm(list = ls())
library(caret)
library(readxl)
source("./dca.R")set.seed(42) #seed
df <- as.data.frame(read.csv(" ")) # original data
dca_results <- dca(data = df,
outcome = "y",
predictors = c("rf", "nn"),
probability = c(TRUE, TRUE))
順番が前後しますが↓
DCAの描画と同じ階層でお願いします↓
dca <- function(data, outcome, predictors, xstart=0.01, xstop=0.99, xby=0.01,
ymin=-0.05, probability=NULL, harm=NULL,graph=TRUE, intervention=FALSE,
interventionper=0.4, smooth=FALSE,loess.span=0.10) {
# LOADING REQUIRED LIBRARIES
require(stats)# data MUST BE A DATA FRAME
if (class(data)!="data.frame") {
stop("Input data must be class data.frame")
}
#ONLY KEEPING COMPLETE CASES
data=data[complete.cases(data[append(outcome,predictors)]),append(outcome,predictors)]# outcome MUST BE CODED AS 0 AND 1
if (max(dataoutcome)>1 | min(dataoutcome)<0) {
stop("outcome cannot be less than 0 or greater than 1")
}
# xstart IS BETWEEN 0 AND 1
if (xstart<0 | xstart>1) {
stop("xstart must lie between 0 and 1")
}
# xstop IS BETWEEN 0 AND 1
if (xstop<0 | xstop>1) {
stop("xstop must lie between 0 and 1")
}
# xby IS BETWEEN 0 AND 1
if (xby<=0 | xby>=1) {
stop("xby must lie between 0 and 1")
}
# xstart IS BEFORE xstop
if (xstart>=xstop) {
stop("xstop must be larger than xstart")
}
#STORING THE NUMBER OF PREDICTORS SPECIFIED
pred.n=length(predictors)
#IF probability SPECIFIED ENSURING THAT EACH PREDICTOR IS INDICATED AS A YES OR NO
if (length(probability)>0 & pred.n!=length(probability)) {
stop("Number of probabilities specified must be the same as the number of predictors being checked.")
}
#IF harm SPECIFIED ENSURING THAT EACH PREDICTOR HAS A SPECIFIED HARM
if (length(harm)>0 & pred.n!=length(harm)) {
stop("Number of harms specified must be the same as the number of predictors being checked.")
}
#INITIALIZING DEFAULT VALUES FOR PROBABILITES AND HARMS IF NOT SPECIFIED
if (length(harm)==0) {
harm=rep(0,pred.n)
}
if (length(probability)==0) {
probability=rep(TRUE,pred.n)
}
#CHECKING THAT EACH probability ELEMENT IS EQUAL TO YES OR NO,
#AND CHECKING THAT PROBABILITIES ARE BETWEEN 0 and 1
#IF NOT A PROB THEN CONVERTING WITH A LOGISTIC REGRESSION
for(m in 1:pred.n) {
if (probability[m]!=TRUE & probability[m]!=FALSE) {
stop("Each element of probability vector must be TRUE or FALSE")
}
if (probability[m]==TRUE & (max(data[predictors[m]])>1 | min(data[predictors[m]])<0)) {
stop(paste(predictors[m],"must be between 0 and 1 OR sepcified as a non-probability in the probability option",sep=" "))
}
if(probability[m]==FALSE) {
model=NULL
pred=NULL
model=glm(data.matrix(data[outcome]) ~ data.matrix(data[predictors[m]]), family=binomial("logit"))
pred=data.frame(model$fitted.values)
pred=data.frame(pred)
names(pred)=predictors[m]
data=cbind(data[names(data)!=predictors[m]],pred)
print(paste(predictors[m],"converted to a probability with logistic regression. Due to linearity assumption, miscalibration may occur.",sep=" "))
}
}# THE PREDICTOR NAMES CANNOT BE EQUAL TO all OR none.
if (length(predictors[predictors=="all" | predictors=="none"])) {
stop("Prediction names cannot be equal to all or none.")
}
######### CALCULATING NET BENEFIT #########
N=dim(data)[1]
event.rate=colMeans(data[outcome])
# CREATING DATAFRAME THAT IS ONE LINE PER THRESHOLD PER all AND none STRATEGY
nb=data.frame(seq(from=xstart, to=xstop, by=xby))
names(nb)="threshold"
interv=nb
nb["all"]=event.rate - (1-event.rate)*nb$threshold/(1-nb$threshold)
nb["none"]=0
# CYCLING THROUGH EACH PREDICTOR AND CALCULATING NET BENEFIT
for(m in 1:pred.n){
for(t in 1:length(nb$threshold)){
# COUNTING TRUE POSITIVES AT EACH THRESHOLD
tp=mean(data[datapredictors[m]>=nb$threshold[t],outcome])*sum(datapredictors[m]>=nb$threshold[t])
# COUNTING FALSE POSITIVES AT EACH THRESHOLD
fp=(1-mean(data[datapredictors[m]>=nb$threshold[t],outcome]))*sum(datapredictors[m]>=nb$threshold[t])
#setting TP and FP to 0 if no observations meet threshold prob.
if (sum(datapredictors[m]>=nb$threshold[t])==0) {
tp=0
fp=0
}
# CALCULATING NET BENEFIT
nb[t,predictors[m]]=tp/N - fp/N*(nb$threshold[t]/(1-nb$threshold[t])) - harm[m]
}
interv[predictors[m]]=(nb[predictors[m]] - nb["all"])*interventionper/(interv$threshold/(1-interv$threshold))
}
# CYCLING THROUGH EACH PREDICTOR AND SMOOTH NET BENEFIT AND INTERVENTIONS AVOIDED
for(m in 1:pred.n) {
if (smooth==TRUE){
lws=loess(data.matrix(nb[!is.na(nbpredictors[m]),predictors[m]]) ~ data.matrix(nb[!is.na(nbpredictors[m]),"threshold"]),span=loess.span)
nb[!is.na(nbpredictors[m]),paste(predictors[m],"_sm",sep="")]=lws$fitted
lws=loess(data.matrix(interv[!is.na(nbpredictors[m]),predictors[m]]) ~ data.matrix(interv[!is.na(nbpredictors[m]),"threshold"]),span=loess.span)
interv[!is.na(nbpredictors[m]),paste(predictors[m],"_sm",sep="")]=lws$fitted
}
}
# PLOTTING GRAPH IF REQUESTED
if (graph==TRUE) {
require(graphics)
# PLOTTING INTERVENTIONS AVOIDED IF REQUESTED
if(intervention==TRUE) {
# initialize the legend label, color, and width using the standard specs of the none and all lines
legendlabel <- NULL
legendcolor <- NULL
legendwidth <- NULL
legendpattern <- NULL
#getting maximum number of avoided interventions
ymax=max(interv[predictors],na.rm = TRUE)
#INITIALIZING EMPTY PLOT WITH LABELS
plot(x=nb$threshold, y=nb$all, type="n" ,xlim=c(xstart, xstop), ylim=c(ymin, ymax), xlab="Threshold probability", ylab=paste("Net reduction in interventions per",interventionper,"patients"))
#PLOTTING INTERVENTIONS AVOIDED FOR EACH PREDICTOR
for(m in 1:pred.n) {
if (smooth==TRUE){
lines(interv$threshold,data.matrix(interv[paste(predictors[m],"_sm",sep="")]),col=m,lty=2)
} else {
lines(interv$threshold,data.matrix(interv[predictors[m]]),col=m,lty=2)
}
# adding each model to the legend
legendlabel <- c(legendlabel, predictors[m])
legendcolor <- c(legendcolor, m)
legendwidth <- c(legendwidth, 1)
legendpattern <- c(legendpattern, 2)
}
} else {
# PLOTTING NET BENEFIT IF REQUESTED
# initialize the legend label, color, and width using the standard specs of the none and all lines
legendlabel <- c("None", "All")
legendcolor <- c(17, 8)
legendwidth <- c(2, 2)
legendpattern <- c(1, 1)
#getting maximum net benefit
ymax=max(nb[names(nb)!="threshold"],na.rm = TRUE)
# inializing new benfit plot with treat all option
plot(x=nb$threshold, y=nb$all, type="l", col=8, lwd=1 ,xlim=c(xstart, xstop), ylim=c(ymin, ymax), xlab="Threshold probability", ylab="Net benefit")
# adding treat none option
lines(x=nb$threshold, y=nb$none,lwd=1)
#PLOTTING net benefit FOR EACH PREDICTOR
for(m in 1:pred.n) {
if (smooth==TRUE){
lines(nb$threshold,data.matrix(nb[paste(predictors[m],"_sm",sep="")]),col="1",lty=m)
} else {
lines(nb$threshold,data.matrix(nb[predictors[m]]),col="1",lty=m)
}
# adding each model to the legend
legendlabel <- c(legendlabel, predictors[m])
legendcolor <- c(legendcolor, "1")
legendwidth <- c(legendwidth, 1)
legendpattern <- c(legendpattern, m)
}
}
# then add the legend
legend("topright", legendlabel, cex=0.8, col=legendcolor, lwd=legendwidth, lty=legendpattern)
}
#RETURNING RESULTS
results=list()
results$N=N
results$predictors=data.frame(cbind(predictors,harm,probability))
names(results$predictors)=c("predictor","harm.applied","probability")
results$interventions.avoided.per=interventionper
results$net.benefit=nb
results$interventions.avoided=interv
return(results)
}

こんな感じになりました?
雑な記事ですみません。
画像を正規化するPythonコード↓
Decision Curve Analysis(DCA)について:意思決定曲線分析
どうも、お久しぶりです。
Decision Curve Analysis(DCA)
今日は、DCAについて、書いてみたいと思います。DCAは予測モデルを評価する際に、使用するものです。
予測モデルの精度には、C統計量やNRI、IDIがありますよね。
雑なRコード↓
今回は、
Decision Curve Analysis(DCA)です。
あまり日本語の論文で使用されていない?のでしょうか。みたことがありません。(もっと論文を読めってことですよね。すみません)
DCAですが、癌の診断の領域では使用されているようです。
DCA:導入(例)
ある病気の検査を受けようか、受けまいか迷っている患者がいます。しかし、病気の詳細な検査Aは体に侵襲をともなうもので強い痛みを伴う可能性があります(例えば生検)。
そこで、まずは簡単で優しいスクリーニング検査Bを受けて、その結果で、ある病気の確率が高ければ、疼痛を伴うけど、詳しい検査をしてみようと考えるものですよね。
そして、簡単な検査Bをした結果、病気である確率が90%でした。
→ この場合は、詳細な検査を実施して、本当に病気であるかどうかをする必要がありそうです。(90%というのは、なんとなくですが)
では、簡単な検査Bで、病気である確率が5%だったらどうでしょうか?
→ 5%でも詳細な検査をするという人もいれば、しないと選択する人もいますよね。
これは、ある病気によって違うかと思います。
例えば、癌の場合はどうでしょうか? 癌の場合、スクリーニング検査で見逃す重さと、過診断して検査をするのとでは、同じ間違いでも重さが違いますよね。
そこで、登場したのが DCAです。DCAは予測モデルによる純利益を考慮して、予測モデルの比較を実施していく手法だそうです。ちなみに、DCAのような予測モデルの純利益(メリット)を考慮した方法は、予後予測の報告のガイドラインで推奨されています。↓↓
https://www.tripod-statement.org/TRIPOD/Translation-Policy
癌の分野では、早期発見が非常に重要なので、癌の論文がDCAの例として扱われることが多いです。あまり、例を出しすぎると(引用しすぎると)、著作権しんがいとなってしまうので、DCAの読み方に関する論文を載せますね↓↓
もう一つ
上記の論文を是非御覧ください。
DCAに関する論文はなんとなく理解しづらいですね。
一応、統計モデルと機械学習モデルでDCAを作成してみました。
(データは適当に作ったサンプルですが)
Decision Curve Analysis(DCA)↓

DCAのコードはまた、今度、公開しますね!!
Rで解析しました。
DCAのRコード↓
画像をヒストグラムにしてみた。Python OpenCV
どうも、体調不良ぎみのGuchiです。
今日は、画像をヒストグラムにしてみましたので更新します。
Pythonで画像をヒストグラムにする
その前に、ヒストグラムは何かご存知でしょうか?Wikipediaをご覧下さい.w
統計では、正規分布やらノンパラやら言われるあんな感じです。雑な感じですみません。
画像といってもカラー画像とグレースケール画像があります。カラー画像は赤青緑の3つの色がそれぞれ合わさって1つの色を表しています。
そして、それぞれ0から255までの256パターンあります。例えば、白だと(255.255.255)になります。
グレースケール画像は単純に0から255までの256パターンです。0が黒、255が白になります。
画像からそれぞれの色の配置がどれくらいなのかを見る事ができます!
では、こちらの画像をヒストグラムにしてみましょう!


次はこちらの画像をヒストグラムにしました。

ヒストグラム(極端ですね、すみません)

カラー写真のヒストグラム コード:
OpenCVはインポートしておいてください
ImgDIR =r"パス"
import cv2
import matplotlib.pyplot as plt
%matplotlib inline
import glob#R, G, Bヒストグラムの表示
def show_imghys(path):# 画像の読み込み
img = cv2.imread(path)
b = img[:,:,0]
g = img[:,:,1]
r = img[:,:,2]
# ヒストグラムの取得
hist_b = cv2.calcHist([b],[0],None,[256],[0,256])
hist_g = cv2.calcHist([g],[0],None,[256],[0,256])
hist_r = cv2.calcHist([r],[0],None,[256],[0,256])
# ヒストグラムの表示
plt.title(path)
plt.plot(hist_r, color='r', label="R",ls=":")
plt.plot(hist_g, color='g', label="G",ls="--")
plt.plot(hist_b, color='b', label="B",ls="-")
#plt.xlim(0, 256)
#plt.ylim(0)plt.legend()
plt.show()
return hist_r,hist_g, hist_b# フォルダ内の画像のパスを取得し、ヒストグラムを表示
ImagePaths = glob.glob(ImgDIR+"\*")
for ImagePath in ImagePaths:
show_imghys(ImagePath)
雑な記事ですみません
κ係数と重み付きκ係数(wカッパ):重み付きカッパはEZRで出来ないのでPythonでしてみた。
どうも。新居に引っ越ししたのですが、◯条工務店に出入りしているGuです。
いきなりのカミングアウトすみません。
κ係数と重み付きκ係数(wカッパ):重み付きカッパはEZRで出来ないのでPythonでしてみた。
(まず、タイトル長い。)
kappa(κ)係数は、順序尺度の検査の一致度をみるものです。
理学療法や作業療法だと、MAS(Modified Ashworth Scale)が有名でしょうか?
κ係数と重み付きκ係数の違いを文字で復習します。
MASを評価する場合、1と4って間違えます??
普通は間違えないですよね?
でも、1と1+であれば、間違えそうじゃないですか?
κ係数の場合は、1と4を間違えても、1と1+を間違えても同じように下がります。でも、1と4を間違える重さと、1と1+を間違える重さって違いますよね?
ということで、重み付きκ係数の登場です。1と4では、重み付きκ係数は下がりますが、1と1+だとあまり下がらないというのが重み付きκ係数です。
ということは、感覚的にですが、κ係数が0.3くらいで、重み付きκ係数が0.8の検査だと、完全に一致はしないかもしれないが、そこそこ近い値となるという感じで解釈しています。
Rコマンダーでκ係数を計算
非常に簡単です。(インポートして、κ係数を押すだなので、解説してもしかたがありません。一応、以前の記事を載せておくので参考にしてください。)
重み付きκ係数はEZRも改変Rコマンダーも出来なかったのでPythonでしてみました。
使用するデータはこちら↓。
検査者AとBが入っていますね。
先に完成形の写真を載せます。

正味、これをコピペするだけです。
scikit-learnはインストールしておいてください。
import pandas as pd
from sklearn.metrics import confusion_matrix, cohen_kappa_score
y_true = df["A"]
y_pred = df["B"]
print("#############Confusion Matrix#############")
conmat = confusion_matrix(y_true, y_pred)
print("RAW: y_true, COLUMN: y_label")
print(conmat)
print("##########################################\n")
k = cohen_kappa_score(y_true, y_pred, weights='quadratic')
print("Weighted Kappa k: {:.3f}".format(k))
これを実行すると、重み付きκ係数がでます。こんな感じにでます↓
Weighted Kappa k: 0.695
よければ、ダウンロードもあります↓
少し難しいかもしれませんが、解説サイトもありました。
https://www.stats-guild.com/analytics/5396
RコマンダーでBland-Altman analysis (ブランドアルトマン分析)
どうも、ちょっと復習がてらに書きます。
Bland-Altman analysis (ブランドアルトマン分析)
Bland-Altman analysisですが、PT協会にも解説がありました↓。
Bland-Altman analysis (ブランドアルトマン分析)は、系統誤差を確認する方法です。
そして、系統誤差は固定誤差と比例誤差に分けられます。
・固定誤差とは真の値に関わらず、特定方向に一定の幅で生じる乖離のことです。例えば、関節可動域測定で1回目の測定より、2回目の測定の方が大きくなってしまうなどが固定誤差です。
・比例誤差とは真の値に比例して増減する、特定方向に生じる誤差のことです。例えば、10m歩行時間を計測する際に、平均5秒で歩く人と平均30秒で歩く人がいた場合、平均30秒で歩く人の方が誤差が大きい場合などです。
固定誤差の算出方法の例
1回目の計測ー2回目の計測の差の95%信頼区間が0を通過する場合、1回目と2回目の計測の差がないということなので、固定誤差がないと判断することが多いようです。
比例誤差の算出方法の例
"1回目の測定と2回目の測定値の差の平均"と"1回目の測定と2回目の測定値の平均"の単回帰分析の結果が有意である場合に比例誤差があると判断することが多いようです。
少し、難しいですが、こちらの論文が詳しく日本語で載っております。
RコマンダーでBland-Altman analysis (ブランドアルトマン分析)
(Rコマンダーのインストールはこちらで↓↓Windowsのみですが、Mac版はこの記事の1番下にインストール方法を記載しています。)
今回はサンプルデータを使用したいと思います。
ダウンロードはこちらから。
OTAとOTBのデータがありますので、コピーしてください。
Rコマンダー⇒データインポート⇒OKの順にお願いします。

次は、統計量⇒信頼性係数⇒Bland-Altman プロットと検定⇒2つの測定値を選択⇒OK
このような画面が出てきたら成功です。

図↓

UIは微妙ですが、無料で使えるソフトなので良いですね。
検査、評価の信頼性についての過去の記事↓
EZR・RコマンダーをMacで使えるようにしてみた!! - 人工知能・リハビリ・日記・理学療法
PythonでぐるなびAPIを使って、飲食店の情報を集める。
どうも、Guchiです。
Pythonで、ぐるなびAPIを使って、飲食店の情報を集める。
社会医学の研究に使用したいと考えております。
特定の住所の飲食店の住所、電話番号をcsvファイルに自動で保存するというものです。
# coding: utf-8
import csv
import requests
import json
import urllib####################
#都道府県名, 市町村名
pref_name = "◯◯県"
city_name = "◯◯市"#出力ファイル名
output_file_name = "guchhi.csv"#ヘッダー行の有無
header_flag = True
####################header = '店名', '住所', '電話番号', '電子マネー決済の可否'
pref_codes = {'北海道': 'PREF01', '青森県': 'PREF02', '岩手県': 'PREF03', '宮城県': 'PREF04', '秋田県': 'PREF05', '山形県': 'PREF06', '福島県': 'PREF07', '茨城県': 'PREF08', '栃木県': 'PREF09', '群馬県': 'PREF10', '埼玉県': 'PREF11', '千葉県': 'PREF12', '東京都': 'PREF13', '神奈川県': 'PREF14', '新潟県': 'PREF15', '富山県': 'PREF16', '石川県': 'PREF17', '福井県': 'PREF18', '山梨県': 'PREF19', '長野県': 'PREF20', '岐阜県': 'PREF21', '静岡県': 'PREF22', '愛知県': 'PREF23', '三重県': 'PREF24', '滋賀県': 'PREF25', '京都府': 'PREF26', '大阪府': 'PREF27', '兵庫県': 'PREF28', '奈良県': 'PREF29', '和歌山県': 'PREF30', '鳥取県': 'PREF31', '島根県': 'PREF32', '岡山県': 'PREF33', '広島県': 'PREF34', '山口県': 'PREF35', '徳島県': 'PREF36', '香川県': 'PREF37', '愛媛県': 'PREF38', '高知県': 'PREF39', '福岡県': 'PREF40', '佐賀県': 'PREF41', '長崎県': 'PREF42', '熊本県': 'PREF43', '大分県': 'PREF44', '宮崎県': 'PREF45', '鹿児島県': 'PREF46', '沖縄県': 'PREF47'}
url = 'https://api.gnavi.co.jp/RestSearchAPI/v3/?keyid=' + api_key + '&pref=' + pref_codes[pref_name] + "&freeword=" + urllib.parse.quote(city_name) + "&hit_per_page=100&offset_page="loop_flag = True
page_num = 1
results = []#APIから取得
while(loop_flag):
now_url = url + str(page_num)
# リクエスト
res = requests.get(now_url)
responses = json.loads(res.text)#エラーレスポンス
if('error' in responses):
loop_flag = False#正常動作
else:
for rest in responses["rest"]:
if(pref_name + city_name in rest["address"]):
results.append([rest["name"], rest["address"], rest["tel"], not rest["e_money"]==''])
page_num +=1#ヘッダー行の追加
if(header_flag):
results = header + results#結果の書き込み
with open(output_file_name, 'w') as f:
writer = csv.writer(f)
writer.writerows(results)
ふと、検査器具って何を持って一般的なのかって思いました。
どうも、Gu●◯です。寝違えてしまい首が痛いです。
いきなりのカミングアウトすみません。(今回はいつも以上に適当な記事です)
リハビリ関連の職種であれば、常に持ち歩いています??
正直、全て持ち歩いている人います??ポケットが重くなります。
近年は、このような便利な器具も出てきています。
ですけど、正味、このアプリで良くないですか?
他にも
スマホ一台で出来ますし。病院でしか勤務したことがない方からは反発されそうです。
今回紹介したアプリ意外にも山のように便利アプリがあるので、検索して見てください。









